Préparation des données
Le but de cette publication est de travailler avec les données de la Covid-19 que nous avons pu récupérer sur le site de l’Institut national de la santé publique du Québec.
Source : INSPQ - Comparaisons provinciales et internationales
Nous souhaitons importer les données dans R et effectuer un traitement graphique de celles-ci.
Nous utiliserons, entre autres, les librairies de tidyverse et ggplot pour se faire.
Procédons avec l’analyse de la 2e vague de la Covid, soit l’analyse de la situation il y a 1 an.
Il faut bien noter que nous travaillons avec des données par 1 000 000 habitants des régions et les nombres de cas et décès fournis par l’INSPQ sont des moyennes mobiles sur 7 jours.
# Tri des dates - Analyse du 23 août 2020 au 20 mars 2021 inclusivement (2e vague)
date_debut <- "2020-08-23" # Jour du début de l'analyse
date_fin <- "2021-03-20" # Jour de fin de l'analyse
# Importer les données de population par province
pop <- read.table("pop_provinces.txt", header = TRUE, sep = "\t") %>%
pivot_wider(names_from = name, values_from = pop) %>%
mutate(PR = AB + MB + SK,
AT = NL + NB + NS + PE)
# Importer les données brutes, calcul des taux des régions regroupées et transformation en table allongée pour les CAS
COVID <- read.table("20211121_Cas_quotidiens_confimes.txt", header = TRUE, sep = "\t") %>%
mutate(Date = as.Date(Date)) %>%# Correction de format des dates
filter(Date >= date_debut & Date <= date_fin) %>%
mutate(PR = round((AB * pop$AB + MB * pop$MB +SK * pop$SK) / pop$PR,2), # Calcul des taux par 100 000 (pondérés) pour les régions des Prairies et Atlantique
AT = round((NL * pop$NL + NB * pop$NB +NS * pop$NS + PE * pop$PE) / pop$AT, 2)) %>%
mutate(Canada = NULL,
NS = NULL, NL = NULL, NB = NULL, PE = NULL,
AB = NULL, MB = NULL, SK = NULL) %>%
pivot_longer(cols = c("AT", "BC", "ON", "PR", "QC"), names_to = "Region", values_to = "Cas") %>%
mutate(Cas = Cas*10) # Transformation par 1M habitants, pour avoir la même échelle que les décès
# Ajout des Décès
COVID <- read.table("20211121_Deces_quotidiens_confimes.txt", header = TRUE, sep = "\t") %>%
mutate(Date = as.Date(Date)) %>%# Correction de format des dates
filter(Date >= date_debut & Date <= date_fin) %>%
mutate(PR = round((AB * pop$AB + MB * pop$MB +SK * pop$SK) / pop$PR,2), # Calcul des taux par 1 000 000 (pondérés) pour les régions des Prairies et Atlantique
AT = round((NL * pop$NL + NB * pop$NB +NS * pop$NS + PE * pop$PE) / pop$AT, 2)) %>%
mutate(Canada = NULL,
NS = NULL, NL = NULL, NB = NULL, PE = NULL,
AB = NULL, MB = NULL, SK = NULL) %>%
pivot_longer(cols = c("AT", "BC", "ON", "PR", "QC"), names_to = "Region", values_to = "Deces") %>%
mutate(COVID, .,
Region = as.factor(Region))
TAUX <- COVID %>%
pivot_longer(cols = c("Cas","Deces"), names_to = "Type", values_to = "TPM") %>% # TPM : Taux par million
mutate(Type = as.factor(Type))
Maintenant que les données sont importées, nous pouvons valider leurs formats et contenus.
# Validation du type de jeux de données
contents(COVID)
## Data frame:COVID 1050 observations and 4 variables Maximum # NAs:0
##
## Levels Class Storage
## Date Date double
## Region 5 integer
## Cas double
## Deces double
##
## +--------+--------------+
## |Variable|Levels |
## +--------+--------------+
## | Region |AT,BC,ON,PR,QC|
## +--------+--------------+
contents(TAUX)
## Data frame:TAUX 2100 observations and 4 variables Maximum # NAs:0
##
## Levels Class Storage
## Date Date double
## Region 5 integer
## Type 2 integer
## TPM double
##
## +--------+--------------+
## |Variable|Levels |
## +--------+--------------+
## | Region |AT,BC,ON,PR,QC|
## +--------+--------------+
## | Type |Cas,Deces |
## +--------+--------------+
# Validation du nombre de données par région
table(TAUX$Region, TAUX$Type)
## Cas Deces
## AT 210 210
## BC 210 210
## ON 210 210
## PR 210 210
## QC 210 210
Nous pouvons voir que nous couvrons bien toute la période de la 2e vague.
Tableaux résumés des cas et décès
Nous pouvons aussi réaliser un regroupement par région et pour le pays complet, de manière à faire ressortir le nombre de cas et de décès confirmés lors de la 2e vague.
Total_Region <- COVID %>%
group_by(Region)%>%
summarise_at(c("Cas","Deces"), sum)%>%
mutate( Cas = round(Cas,0),
Deces = round(Deces,0))
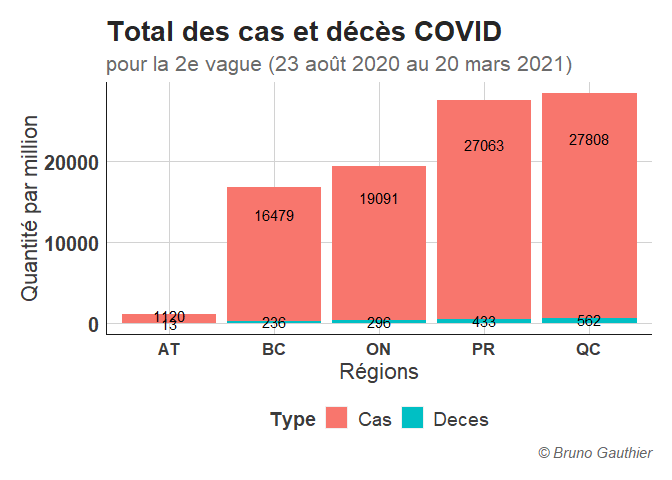
Total_Region
## Region Cas Deces
## 1 AT 1120 13
## 2 BC 16479 236
## 3 ON 19091 296
## 4 PR 27063 433
## 5 QC 27808 562
Total_Pays <- COVID %>%
summarise_at(c("Cas","Deces"), sum) %>%
mutate( Cas = round(Cas,0),
Deces = round(Deces,0))
Total_Pays
## Cas Deces
## 91563 1541
Graphiques
L’analyse graphique nous permettra de mieux comparer les données entres elles.
Nombre de cas et de décès total par région
Total_Region %>%
pivot_longer(cols = c("Cas","Deces"), names_to = "Type", values_to = "TPM") %>%
mutate(Type = as.factor(Type)) %>%
ggplot(aes(x = Region, y = TPM, fill = Type, label = TPM))+
geom_bar(stat = "identity")+
theme+
labs( x = "Régions",
y = "Quantité par million",
title = "Total des cas et décès COVID",
subtitle = "pour la 2e vague (23 août 2020 au 20 mars 2021)",
caption = "© Bruno Gauthier") +
geom_text(position = position_stack(vjust = 0.8))
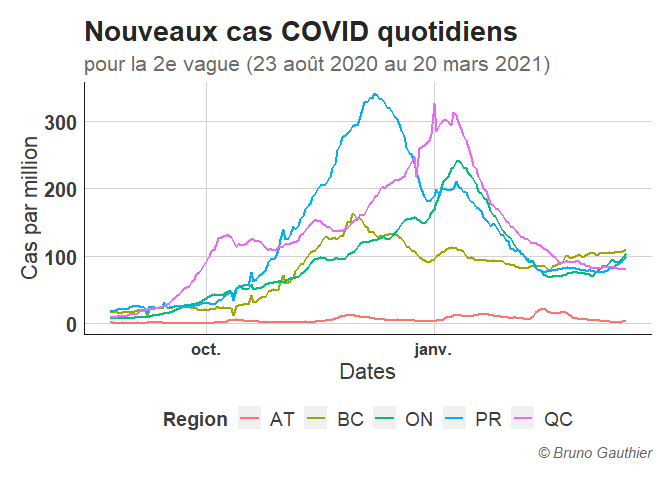
Évolution des cas et des décès dans le temps
ggplot(COVID, aes(x=Date, y=Cas, color = Region)) +
geom_line(size = 1)+
theme+
labs( x = "Dates",
y = "Cas par million",
title = "Nouveaux cas COVID quotidiens",
subtitle = "pour la 2e vague (23 août 2020 au 20 mars 2021)",
caption = "© Bruno Gauthier")
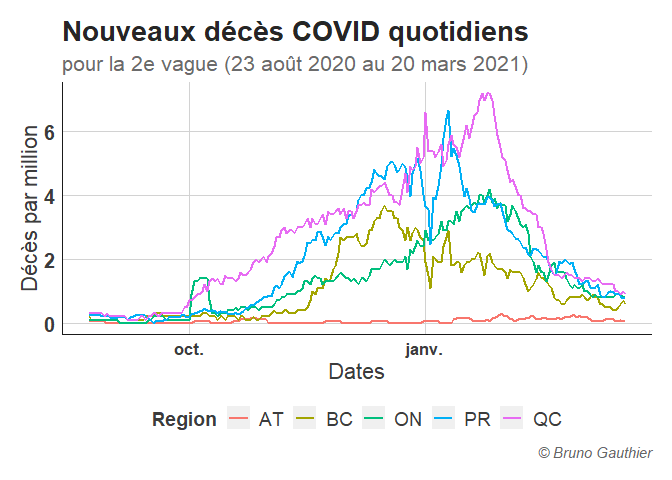
ggplot(COVID, aes(x=Date, y=Deces, color = Region)) +
geom_line(size = 1)+
theme+
labs( x = "Dates",
y = "Décès par million",
title = "Nouveaux décès COVID quotidiens",
subtitle = "pour la 2e vague (23 août 2020 au 20 mars 2021)",
caption = "© Bruno Gauthier")
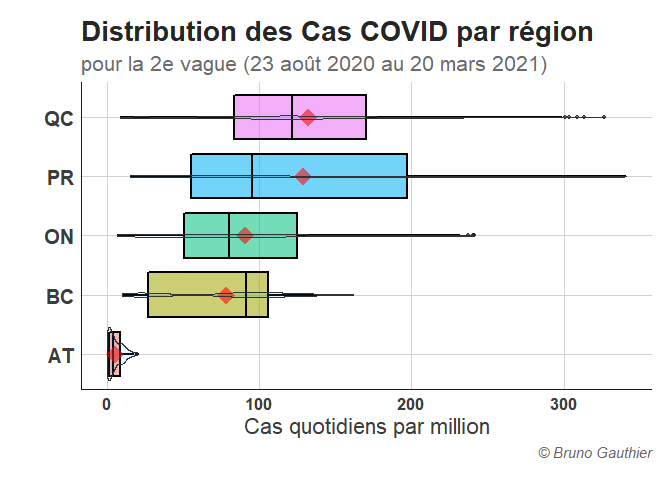
Distribution des cas et décès quotiens par région
boxplot_cas <- COVID %>%
ggplot(aes(y=Region, x=Cas, fill = Region, alpha = 0.9))+
geom_boxplot(outlier.size = 1.7,
outlier.shape = 20,
lwd = 0.8,
fatten = 1.2,
col = "black")+
stat_summary(fun=mean, geom="point", shape=18, size=6, color="red", fill="red")+
geom_violin(alpha = 0.1,
fill = "deepskyblue2")+
theme+
labs(title = 'Distribution des Cas COVID par région',
subtitle = 'pour la 2e vague (23 août 2020 au 20 mars 2021)',
y = "",
x= "Cas quotidiens par million",
fill = "",
caption = "© Bruno Gauthier")+
theme(legend.position = "none")
boxplot_cas
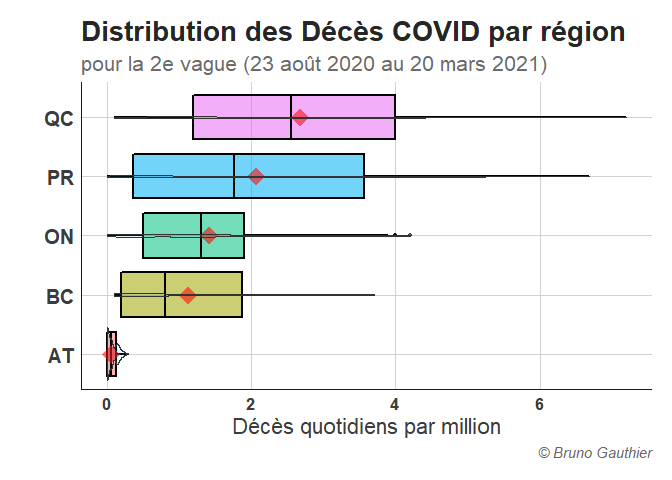
boxplot_deces <- COVID %>%
ggplot(aes(y=Region, x=Deces, fill = Region, alpha = 0.9))+
geom_boxplot(outlier.size = 1.7,
outlier.shape = 20,
lwd = 0.8,
fatten = 1.2,
col = "black")+
stat_summary(fun=mean, geom="point", shape=18, size=6, color="red", fill="red")+
geom_violin(alpha = 0.1,
fill = "deepskyblue2")+
theme+
labs(title = 'Distribution des Décès COVID par région',
subtitle = 'pour la 2e vague (23 août 2020 au 20 mars 2021)',
y = "",
x= "Décès quotidiens par million",
fill = "",
caption = "© Bruno Gauthier")+
theme(legend.position = "none")
boxplot_deces
La suite
Dans une 2e partie à venir, nous nous intéresserons à l’analyse statistique de ces données, en particulier pour le Québec.